CD630_20810 xdhC1
Summary
| Title | Product | Short Name | Synonyms | Start | End | Strand | Sequence Name | Essentiality (Tn-Seq) | Essentiality (in vivo) | Essentiality (Nutrient-rich broth) |
|---|---|---|---|---|---|---|---|---|---|---|
| CD630_20810 | Xanthine dehydrogenase iron-sulfur bindingsubunit XdhC1 | xdhC1 | 2401687 | 2402133 | - | Chromosome |
No |
No |
No |
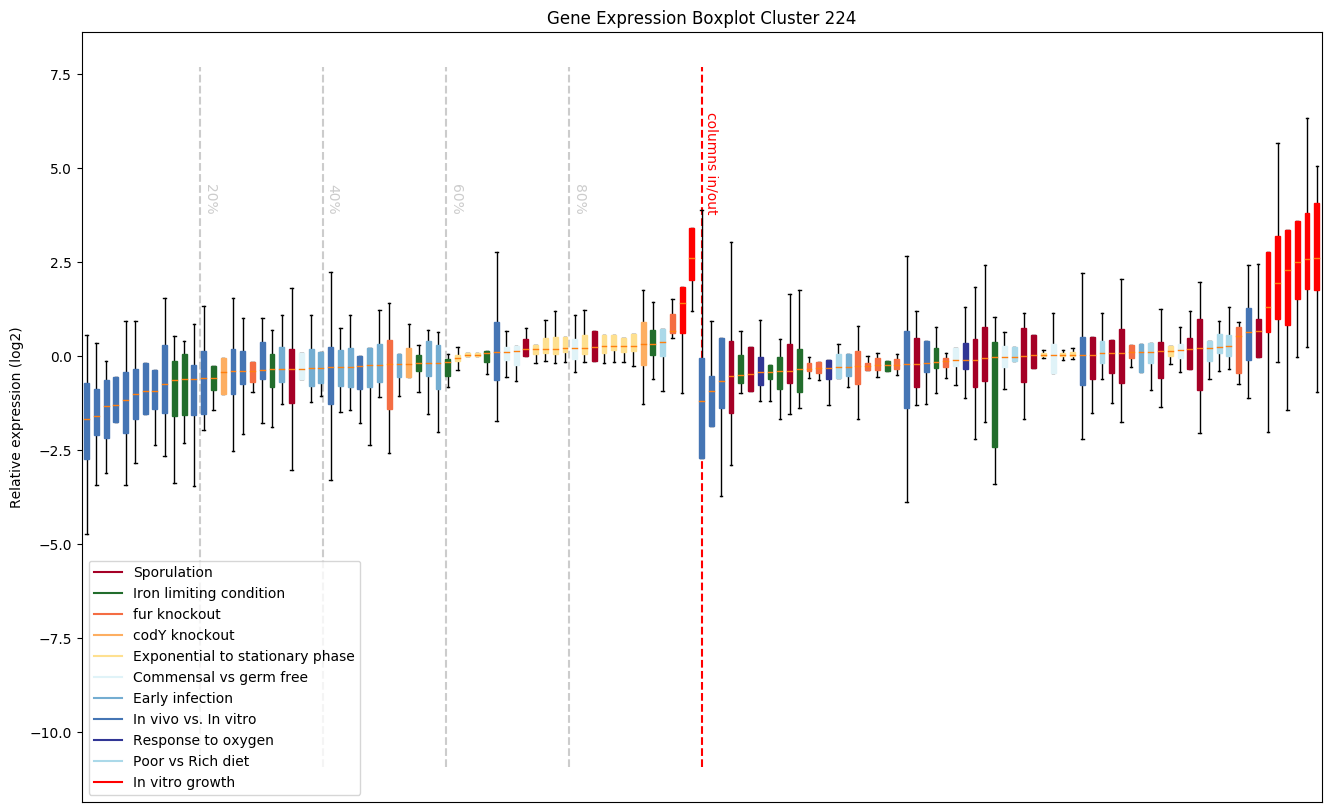
Expression profile of the gene across conditions
|
|
CD630_20810 is a member of the following modules
Regulatory module refers to set of genes that are conditionally co-regulated under subset of the conditions. Identification of modules integrates co-expression, de-novo motif identification, and other functional associations such as operon information and protein-protein interactions. Modules are based on cMonkey algorithm.
Motifs: De novo predicted motifs for each module are listed in the module page as motif logo images along with associated prediction statistics (e-values).
* Given that translation start was used to define the 3' end of putative promoters, some cis-regulatory motifs are likely to be ribosome binding sites (i.e., Shine-Dalgarno sequence). RBS is typically a six nucleotide sequence 5'-GGAGGA-3'.
| Module | Gene Count | Motif 1 e-value | Motif 1 logo | Residual | Expression | TF Hypergeometric | TF Inferelator |
|---|---|---|---|---|---|---|---|
| 61 | 23 | 0.47 |
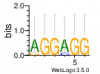 |
0.47 |
 |
NA | CD630_02760 |
| 224 | 26 | 0.17 |
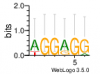 |
0.49 |
 |
NA | CD630_02760, CD630_32140 |
Metabolic Reactions
| Desc | Reaction | GPR | Proteins | Subsystem | |
|---|---|---|---|---|---|
| ID_350 |
hypoxanthine:NAD+ oxidoreductase |
hxan[c] + nad[c] + h2o[c] -> xan[c] + nadh[c] + h[c] | (CD630_20740) or (CD630_20730) or (CD630_20790 and CD630_20800 and CD630_20810) or (CD630_20870) or (CD630_20880) or (CD630_20990 and CD630_21000 and CD630_21010) or (CD630_31770) | Q188E2, Q188G8, Q188E9, Q188G6, Q188E2, Q188E1, Q188E9, Q188G6, Q188E2, Q188E7, Q188E9, Q188G6, Q188E2, Q188G8, Q188F6, Q188G6, Q188E2, Q188E1, Q188F6, Q188G6, Q188E2, Q188E7, Q188F6, Q188G6, Q188E2, Q188G8, Q188G9, Q188G6, Q188E2, Q188E1, Q188G9, Q188G6, Q188E2, Q188E7, Q188G9, Q188G6, Q188E8, Q188G8, Q188E9, Q188G6, Q188E8, Q188E1, Q188E9, Q188G6, Q188E8, Q188E7, Q188E9, Q188G6, Q188E8, Q188G8, Q188F6, Q188G6, Q188E8, Q188E1, Q188F6, Q188G6, Q188E8, Q188E7, Q188F6, Q188G6, Q188E8, Q188G8, Q188G9, Q188G6, Q188E8, Q188E1, Q188G9, Q188G6, Q188E8, Q188E7, Q188G9, Q188G6, Q181U2, Q188G8, Q188E9, Q188G6, Q181U2, Q188E1, Q188E9, Q188G6, Q181U2, Q188E7, Q188E9, Q188G6, Q181U2, Q188G8, Q188F6, Q188G6, Q181U2, Q188E1, Q188F6, Q188G6, Q181U2, Q188E7, Q188F6, Q188G6, Q181U2, Q188G8, Q188G9, Q188G6, Q181U2, Q188E1, Q188G9, Q188G6, Q181U2, Q188E7, Q188G9, Q188G6, Q188F4, Q188G8, Q188E9, Q188G6, Q188F4, Q188E1, Q188E9, Q188G6, Q188F4, Q188E7, Q188E9, Q188G6, Q188F4, Q188G8, Q188F6, Q188G6, Q188F4, Q188E1, Q188F6, Q188G6, Q188F4, Q188E7, Q188F6, Q188G6, Q188F4, Q188G8, Q188G9, Q188G6, Q188F4, Q188E1, Q188G9, Q188G6, Q188F4, Q188E7, Q188G9, Q188G6 | Purine metabolism |
COG
| COG Category | COG codes | COG Name |
|---|---|---|
| METABOLISM | C | Energy production and conversion |
Other Annotations
| Duplicate Locus | Protein Names |
|---|---|
| No | Xanthine dehydrogenase iron-sulfur binding subunit XdhC1 (EC 1.17.1.4) |
Functional
| Function | Pathway | Protein Families |
|---|---|---|
| NA | NA | NA |
Cross Reference
| EnsemblBacteria ID | KEGG IDs | PATRIC IDs | Uniprot ID |
|---|---|---|---|
| CAJ68966 | cdf:CD630_20810, pdc:CDIF630_02304 | fig|272563.120.peg.2195 | Q188E9 |